数据分析软件
现有的商业SPR系统通常测量传感芯片或光纤尖端的反射强度。 即使芯片上有多个传感点,来自多个点的反射也会被采集为单个累积信号。 对于基于平面金膜的 SPR 系统尤其如此,该系统通常假设对受体-靶标结合具有统一的响应。 然而,当靶标浓度达到阿摩尔水平时,这意味着每个点只有很少的靶标拷贝。 假设整个传感点的折射率变化均匀已不再合适。 因此,必须重新发明数据分析算法来解决这个问题。 由于我们采用 CMOS 阵列传感器作为相机来捕获数据,因此采用像素级处理方式来解决该问题更为合理。
此外,我们的QPLoC生物传感器是通过3D打印制成的,TiN纳米立方体在生物芯片表面的位置相当随机。 因此,不再适合假设微孔的一致响应用于阿摩尔水平的生物传感。 因此,我们将逐像素数据处理例程引入到我们的数据分析软件中。 我们的数据分析例程的逻辑相当简单:
- 将实验的所有帧按顺序加载到PC工作站内存中,
- 输入待分析微孔的中心,计算微孔内所有像素的纵横坐标(直径100像素的微孔约为8000像素),
- 对于上面的每个像素,提取历史原始数据集,然后使用 B 样条曲线拟合获得每个数据集的低通结果,
- 根据(3)的结果求出相位沿时域变化的最大值和最小值,并确定最大值和最小值的位置,
- 根据(4)计算最大值和最小值之间的相位差,并检查最大值和最小值之间的间隔是否足够。 如果间隔太短,则由于像素不稳定,计算出的相位差无效。 否则,相位差有效并保留,
- 对微孔内的所有像素重复 (3) 至 (4),并为具有有效相位变化的所有像素重建 2D 相位图。 对于相位数据无效的像素,将相位值调整为零,
- 根据重建的二维相位图,计算平均值和标准差来表示生物传感性能。
由于每个原始帧消耗约20Mbytes的内存,并且每个实验有800帧,因此需要16Gbytes的内存来存储所有原始帧。 当然,对于持续时间较长的实验,需要额外的内存。 使用现代 PC 工作站,每个微孔的像素计算速度非常快,处理 800 帧只需不到一秒。 我们的系统目前使用开源分析例程库,包括 OpenCV [1]、GNU Scientific Library [2],图形用户界面是在小型企业许可证下使用 Qt® [3] 开发的。
我们的分析软件的典型结果如下。 图 1 显示了特定微孔处的相图。 它是阿摩尔浓度的 CRP 阳性血清。 相位图的平均值约为0.20弧度。 而图2显示了相同微孔中CRP阴性血清在相同次数的重复稀释下的相图。 平均值仅为0.07弧度。
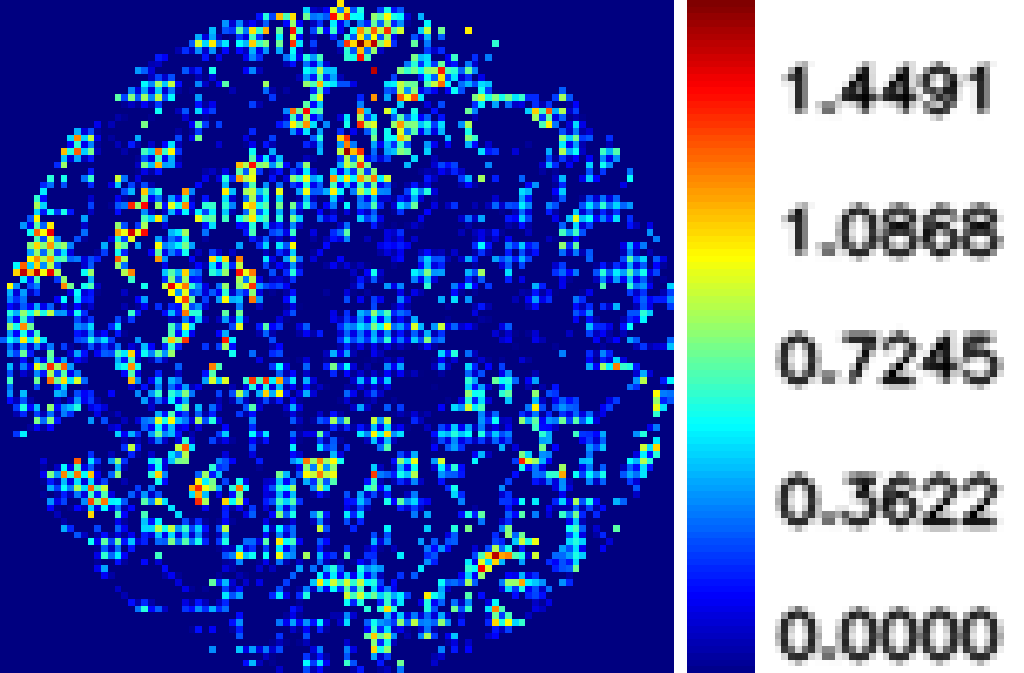
CRP 阳性血清平均值为 0.20 弧度
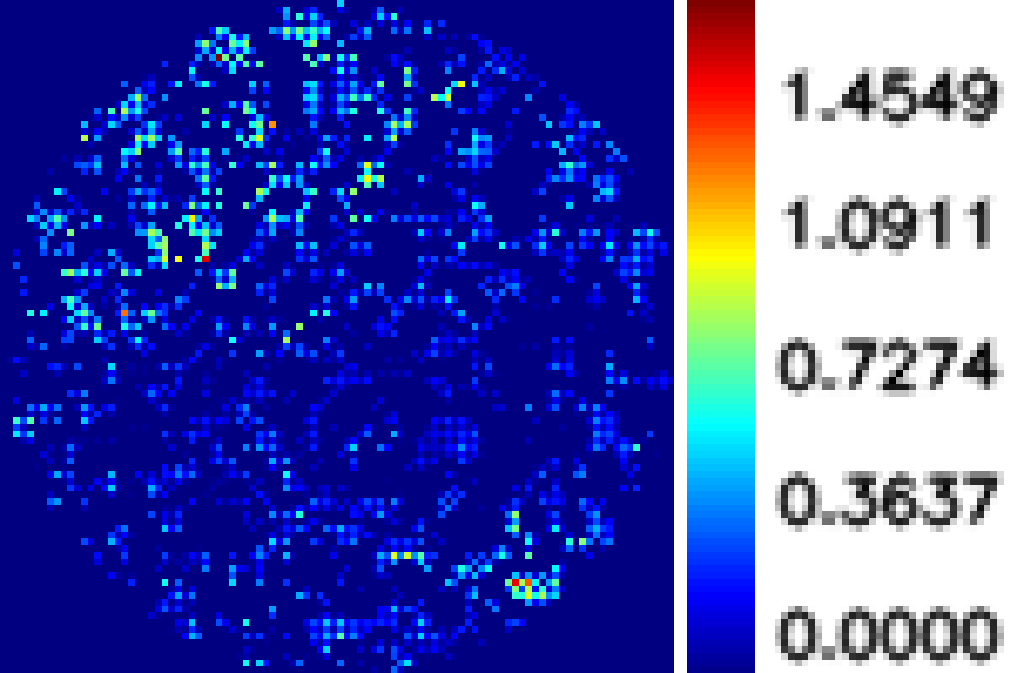
CRP 阴性血清平均值为 0.07 弧度
参考链接
[1] OpenCV, https://opencv.org/
[2] GNU Scientifc Library, https://www.gnu.org/software/gsl/
[3] Qt, https://www.qt.io/